Contexte
Globules blancs et recombinaisons VDJ
Les globules blancs jouent un rôle clé dans l'immunité. Les lymphocytes B et T sont adaptées aux différentes infections, produisant des anticorps et des récepteurs spécifiques à tel ou tel virus ou bactérie. Pour cela, une partie de la séquence d'ADN des globules blancs est transformé par la recombinaison VDJ, recombinaison donnant des milliards de possibilités différentes à partir d'un répertoire de quelques gènes.
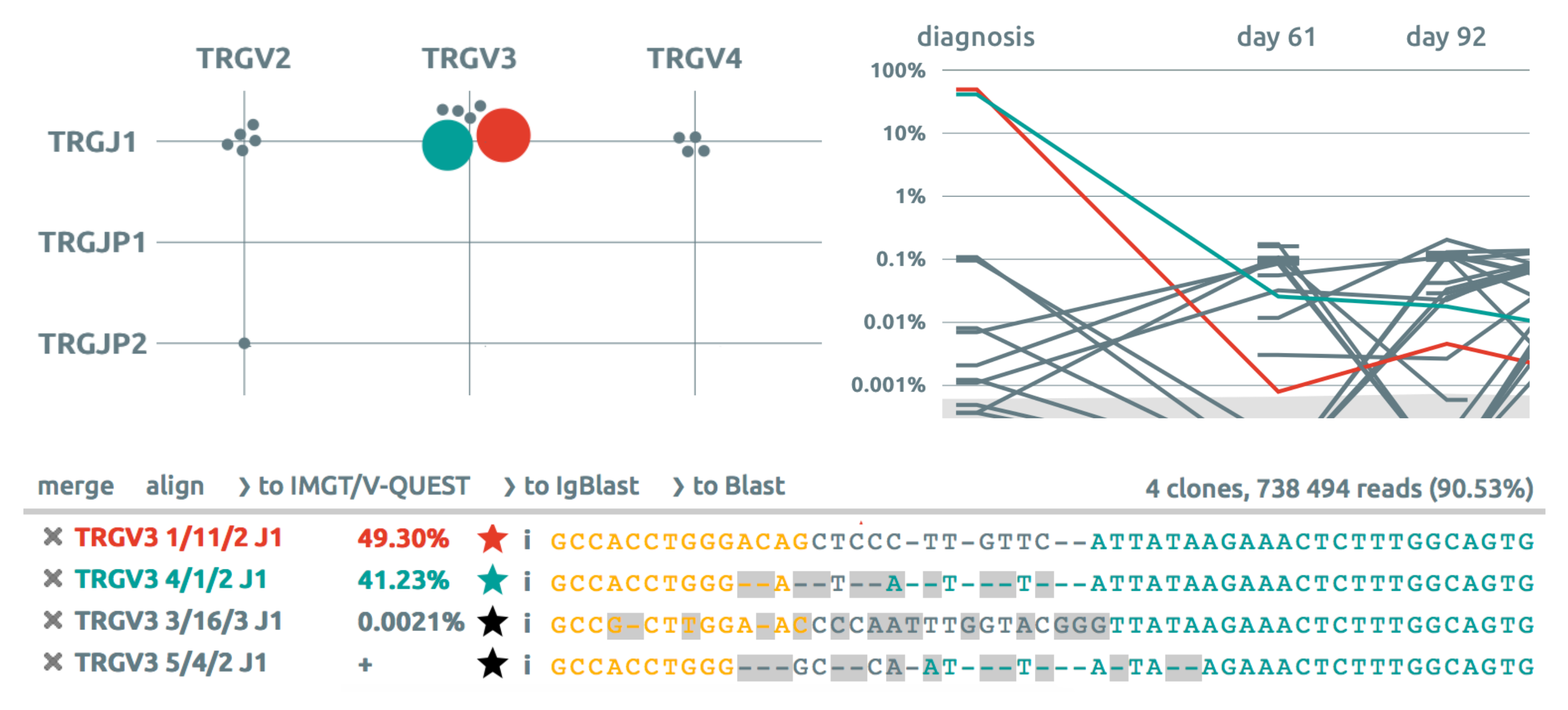
Aujourd'hui, grâce aux progrès colossaux réalisés dans le domaine du séquençage de l'ADN via les séquenceurs à haut débit, on peut déterminer la séquence ADN d'un grand nombre de globules blancs et, grâce aux méthodes RepSeq (Repertoire Sequencing), avoir une vue complète sur une population de recombinaisons V(D)J. Connaître les réarrangements VDJ des globules blancs, c'est connaître avec précion la réponse immunitaire. C'est aussi un outil primordial pour le suivi des leucémies, où l'on évalue la concentration des lymphoblastes possédant le même réarrangement VDJ que celui du diagnostic.
Vidjil et Vidjil-algo
Destinés aux laboratoires effectuant du suivi de leucémie ou des recherches en immunologie, la plateforme Vidjil et l'algorithme Vidjil-algo ont été réalisés en open-source par l'équipe de bioinformatique Bonsai (CRIStAL et Inria Lille) en collaboration avec l'hôpital de Lille et d'autres hôpitaux français et étrangers. Vidjil-algo, implémenté en C++, a pour but de compter les globules blancs en les regroupant en clones suivant leurs séquence d'ADN et leur réarrangement VDJ, en utilisant une heuristique à base de graines. L'application web, développée en Javascript objet et HTML5 (client) et python/web2py (serveur), permet de visualiser des résultats d'analyse comme ceux de Vidjil-algo et de les annoter.
Le serveur public app.vidjil.org a ouvert fin 2014, et plusieurs hôpitaux installent désormais Vidjil et Vidjil-algo dans leurs laboratoires. La plateforme a aujourd'hui plus de 40 laboratoires utilisateurs en France et dans le monde. Sur les deux dernières années, plus de 2 000 échantillons de patients atteints de leucémies aigües ou chroniques ont été ainsi analysés avec l'aide de Vidjil et Vidjil-algo. En janvier 2018, le consortium VidjilNet démarre au sein de la Fondation Inria. Regroupant nos usagers hospitaliers, VidjilNet soutient le développement et le support de Vidjil et de Vidjil-algo. Deux postes d'ingénieur sont en cours de création au sein de ce consortium.
But du stage
Le but de ce stage sera d'améliorer la plateforme Vidjil dans le traitement de résultats provenant de plusieurs logiciels d'analyse. En effet, bien que l'algorithme Vidjil-algo soit au coeur de la plateforme Vidjil, certains utilisateurs souhaitent pouvoir utiliser d'autres méthodes algorithmiques tout en bénéficiant de la facilité de l'application web. Il peut être aussi intéressant de comparer les résultats de Vidjil-algo et ceux d'autres logiciels, ou, plus généralement, de comparer plusieurs réglages de ces logiciels. Des logiciels (MiXCR, IGoR) sont ainsi déjà inclus ou en cours d'inclusion, mais d'autres tels Decombinator, IgReC seraient pertinents à tester.
Concrètement, après une bibliographie sur la recombinaison V(D)J et les logiciels RepSeq, le travail du stage portera ainsi sur trois points:
- tester plusieurs logiciels de RepSeq sur des données de répertoire immunologiques et analyser de manière critique leurs performances (bioanalyse),
- réaliser au moins deux wrappers pour que des logiciels de RepSeq produisent une sortie au format Vidjil (scripts python ou patchs dans les logiciels),
- améliorer la visualisation dans le client web des clones provenant à la fois de Vidjil-algo et d'autres logiciels, notamment par l'ajout d'axes d'analyse et le traitement des avertissements (développement web .js).
Compétences et qualité du code
Compétences souhaitées: connaissances dans les logiciels de bioinformatique, développement agile (git/gitlab, tests, intégration continue), développement Python, développement web javascript, tests unitaires et fonctionnels. Une connaissance en immunologie serait un plus.
Le code sera écrit avec grand soin, documenté et testé. En cas de succès du projet, les développements effectués seront validés par les équipes hospitalières avec lesquelles nous travaillons et déployés pour tous nos utilisateurs.
Possibilités d'emploi
L'équipe Bonsai a régulièrement des propositions de CDD sur ce projet (4 ingénieurs dans l'équipe depuis les trois dernières années), et des postes d'ingénieur en CDI sont en création au sein du consortium VidjilNet.